Phosphoglycérate mutase
| Phosphoglycérate mutase 1 (cérébrale) | ||
 Phosphoglycérate mutase 1 humaine (PDB 1YFK) | ||
| Caractéristiques générales | ||
|---|---|---|
| Symbole | PGAM1 | |
| N° EC | 5.4.2.11 | |
| Homo sapiens | ||
| Locus | 10q24.1 | |
| Masse moléculaire | 28 804 Da[1] | |
| Nombre de résidus | 254 acides aminés[1] | |
| Liens accessibles depuis GeneCards et HUGO. | ||
| Phosphoglycérate mutase 2 (musculaire) | ||
| Caractéristiques générales | ||
| Symbole | PGAM2 | |
| N° EC | 5.4.2.11 | |
| Homo sapiens | ||
| Locus | 7p13 | |
| Masse moléculaire | 28 766 Da[1] | |
| Nombre de résidus | 253 acides aminés[1] | |
| Liens accessibles depuis GeneCards et HUGO. | ||
La phosphoglycérate mutase (PGM) est une isomérase qui catalyse la réaction :
Cette enzyme intervient à la 8e étape de la glycolyse en catalysant le transfert d'un groupe phosphate de l'atome de carbone C-3 vers l'atome C-2 d'une molécule de 3-phospho-D-glycérate (3PG) pour la convertir en 2-phospho-D-glycérate (2PG).
Isoformes dépendante et indépendante du 2,3-bisphosphoglycérate
[modifier | modifier le code]| N° EC | EC |
|---|
| IUBMB | Entrée IUBMB |
|---|---|
| IntEnz | Vue IntEnz |
| BRENDA | Entrée BRENDA |
| KEGG | Entrée KEGG |
| MetaCyc | Voie métabolique |
| PRIAM | Profil |
| PDB | RCSB PDB PDBe PDBj PDBsum |
| N° EC | EC |
|---|---|
| Cofacteur(s) | Mn2+ ou Co2+ |
| IUBMB | Entrée IUBMB |
|---|---|
| IntEnz | Vue IntEnz |
| BRENDA | Entrée BRENDA |
| KEGG | Entrée KEGG |
| MetaCyc | Voie métabolique |
| PRIAM | Profil |
| PDB | RCSB PDB PDBe PDBj PDBsum |
Il existe deux isoformes de phosphoglycérate mutase, dont le mécanisme réactionnel est opposé :
- la phosphoglycérate mutase 2,3-bisphosphoglycérate-dépendante (EC ), présente notamment parmi les vertébrés, les platyhelminthes, les mollusques, les annélides, les crustacés, les insectes, les algues, les mycètes, les levures et certaines bactéries (particulièrement à Gram négatif), est activée par phosphorylation d'un résidu d'histidine à partir du 2,3-bisphosphoglycérate ; ce phosphate est cédé au 3PG pour former le 2,3-BPG, qui cède son autre phosphate à l'enzyme pour donner le 2PG :

|
+ enzyme-phosphate | 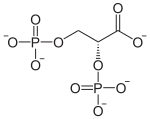
|
+ enzyme enzyme-phosphate + | 
|
| 3PG | 2,3-BPG | 2PG |
- la phosphoglycérate mutase 2,3-bisphosphoglycérate-indépendante (EC ), présente notamment parmi les trachéophytes (plantes vasculaires), les algues, les mycètes, les nématodes, les éponges, les radiaires, les myriapodes, les arachnides, les échinodermes, les archées et certaines bactéries (particulièrement à Gram positif)[4], possède une activité maximum en l'absence de 2,3-bisphosphoglycérate ; elle utilise comme cofacteurs deux cations de manganèse Mn2+ ou, chez certaines espèces, deux cations de cobalt Co2+, qui participent au transfert du groupe phosphate du 3PG sur un résidu de sérine de l'enzyme, qui rend ce phosphate au substrat pour donner du 2PG[3] :

|
+ enzyme enzyme-phosphate + | 
|
enzyme + | 
|
| 3PG | Glycérate | 2PG |
Réactions catalysées
[modifier | modifier le code]Une phosphoglycérate mutase est capable de catalyser trois réactions différentes :
- une réaction de type mutase consistant en l'interconversion du 3-phosphoglycérate en 2-phosphoglycérate[5],[6] ;
- une réaction de type phosphatase convertissant le 2,3-bisphosphoglycérate en phosphoglycérate[7],[8] ;
- une activité synthase convertissant le 2,3-bisphosphoglycérate en 1,3-bisphosphoglycérate de manière semblable à la bisphosphoglycérate mutase. Des études cinétiques et structurelles ont montré que la phosphoglycérate mutase et la bisphosphoglycérate mutase sont paralogues[7]. Ces deux enzymes appartiennent à la même superfamille que le domaine phosphatase de la phosphofructokinase-2 et de la phosphatase acide prostatique (en)[9].
Mécanisme de la phosphoglycérate mutase 2,3-bisphosphoglycérate-dépendante
[modifier | modifier le code]L'activité mutase implique deux groupes phosphate distincts, de sorte que le groupe phosphate du carbone C-2 du produit de la réaction n'est pas le même que celui du carbone C-3 du substrat. Le site actif de l'enzyme à l'état initial contient un résidu de phosphohistidine issu de la phosphorylation d'un résidu d'histidine[10]. Lorsque le 3-phosphoglycérate entre dans le site actif, la phosphohistidine est positionnée de façon à faciliter le transfert de son groupe phosphate au C-2 du substrat pour former l'intermédiaire 2,3-bisphosphoglycérate. La déphosphorylation du résidu de phosphohistidine déclenche une modification conformationnelle de l'enzyme qui place le groupe phosphate en C-3 en face du résidu d'histidine déphosphorylé : ce dernier est rephosphorylé, ce qui convertir le 2,3-bisphosphoglycérate en 2-phosphoglycérate, tandis que l'enzyme retrouve sa conformation initiale, qui libère le substrat.
Notes et références
[modifier | modifier le code]- Les valeurs de la masse et du nombre de résidus indiquées ici sont celles du précurseur protéique issu de la traduction du gène, avant modifications post-traductionnelles, et peuvent différer significativement des valeurs correspondantes pour la protéine fonctionnelle.
- (en) S. I. Winn, H. C. Watson, R. N. Harkins et L. A. Fothergill, « Structure and Activity of Phosphoglycerate Mutase », Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences, vol. 293, no 1063, , p. 121-130 (PMID 6115412, DOI 10.1098/rstb.1981.0066, JSTOR 2395663, Bibcode 1981RSPTB.293..121W, lire en ligne)
- (en) Mark J. Jedrzejas, Monica Chander, Peter Setlow et Gunasekaran Krishnasamy, « Mechanism of catalysis of the cofactor-independent phosphoglycerate mutase from Bacillus stearothermophilus. Crystal structure of the complex with 2-phosphoglycerate », Journal of Biological Chemistry, vol. 275, no 30, , p. 23146-23153 (PMID 10764795, DOI 10.1074/jbc.M002544200, lire en ligne)
- (en) Mark J. Jedrzejas, Monica Chander, Peter Setlow et Gunasekaran Krishnasamy, « Structure and mechanism of action of a novel phosphoglycerate mutase from Bacillus stearothermophilus », The EMBO Journal, vol. 19, no 7, , p. 1419-1431 (PMID 10747010, PMCID 313928, DOI 10.1093/emboj/19.7.1419, lire en ligne)
- (en) Ryuzo Sasaki, Shigeru Utsumi, Etsuro Sugimoto et Hideo Chiba, « Subunit Structure and Multifunctional Properties of Yeast Phosphoglyceromutase », The FEBS Journal, vol. 66, no 3, , p. 523-533 (PMID 182494, DOI 10.1111/j.1432-1033.1976.tb10578.x, lire en ligne)
- (en) Z. B. Rose et S. Dube, « Rates of phosphorylation and dephosphorylation of phosphoglycerate mutase and bisphosphoglycerate synthase », Journal of Biological Chemistry, vol. 261, no 16, , p. 4817-4822 (PMID 8447, lire en ligne)
- (en) Z. B. Rose et S. Dube, « Phosphoglycerate mutase. Kinetics and effects of salts on the mutase and bisphosphoglycerate phosphatase activities of the enzyme from chicken breast muscle », Journal of Biological Chemistry, vol. 253, no 23, , p. 8583-8592 (PMID 213437, lire en ligne)
- (en) Ryuzo Sasaki, Masaaki Hirose, Etsuro Sugimoto et Hideo Chiba, « Studies on a role of the 2,3-diphosphoglycerate phosphatase activity in the yeast phosphoglycerate mutase reaction », Biochimica et Biophysica Acta (BBA) - Enzymology, vol. 227, no 3, , p. 595-607 (PMID 4328052, DOI 10.1016/0005-2744(71)90010-6, lire en ligne)
- (en) Yanli Wang, Zhiyi Wei, Lin Liu, Zhongjun Cheng, Yajing Lin, Fengyuan Ji, Weimin Gong, « Crystal structure of human B-type phosphoglycerate mutase bound with citrate », Biochemical and Biophysical Research Communications, vol. 331, no 4, , p. 1207-1215 (PMID 15883004, DOI 10.1016/j.bbrc.2005.03.243, lire en ligne)
- (en) H. G. Britton et J. B. Clarke, « The mechanism of the phosphoglycerate mutase reaction », Biochemical Journal, vol. 112, no 1, , p. 10P-11P (PMID 5774486, PMCID 1187664, DOI 10.1042/bj1120010Pb, lire en ligne)



