DNA mitokondrial

DNA mitokondriala (DNAmt edo mDNA) [1]mitokondrioetan dagoen DNA da. Mitokondrioa zelula eukariotoetan aurkitzen den organulu bat da, zeinak elikagaietatik abiatuz energia kimikoa sortzen duen (ATP moduan, adibidez). DNA mitokondriala zelula eukariotoen DNAren zati txiki bat baino ez da; izan ere, DNAren zatirik handiena zelularen nukleoan dago (DNAn). Landare eta algetan ordea, DNA plastidoetan ere aurkitzen da, hala nola, kloroplastoetan.
Sekuentziatu zen giza genomaren lehen zati garrantzitsuena DNA mitokondriala izan zen.[2] Sekuentziazio honen arabera, DNAmt-ak 16.569 base-pare ditu eta 13 proteina kodetzen ditu.

Animalien DNAmt-ak markatzaile genetiko nuklearrek baino azkarrago eboluzionatzen duenez, [3][4][5] filogenetikaren eta biologia ebolutiboaren funtsezko oinarri da. Populazioen arteko harremana ere azter daiteke; hori dela eta, antropologian eta biogeografian garrantzi handia du.
Jatorria
[aldatu | aldatu iturburu kodea]DNA mitokondrialaren jatorri ebolutiboa berezia da; uste denez, DNAmt-a zelula eukarioto arbasoek irentsitako bakterioen genoma zirkularretik eratorria da. Teoria honi teoria endosinbiotiko deritzo. DNA mitokondrialak (DNAmt) eta DNA nuklearrak (DNAn) kodetzen dituzte mitokondrioko proteinak. Hala ere, mitokondrioko proteinak kodetzen dituen DNAn-ren gene askok jatorria bakterioetan dutela uste da; eboluzioan zehar gene hauek nukleoko genoman txertatzen joan dira.[6]
Genomaren egitura eta dibertsitatea
[aldatu | aldatu iturburu kodea]Organismo guztiak kontuan hartuta, genoma-mitrokondrialaren 6 genoma-mota bereizten dira, sailkapen hori egiteko ezaugarri hauek hartzen dira kontuan: egitura (zirkularra edo lineala), tamaina, plasmidoen antzeko egiturak edo introiak, eta material genetikoa molekula berezi bat edo molekula talde homogeneo edo heterogeno izatea. [7]
| Genoma mota | Erreinua | Introiak | Tamaina | Egitura | Deskribapena |
|---|---|---|---|---|---|
| 1 | Animalia | Ez | 11-28 kbp | Zirkularra | Molekula bakarra |
| 2 | Fungi, Landare, Protista | Bai | 19-1000 kbp | Zirkularra | Molekula bakarra |
| 3 | Fungi, Landare, Protista | Ez | 20-1000 kbp | Zirkularra | Molekula erraldoia plasmido moduko egitura txikiekin |
| 4 | Protista | Ez | 1-200 kbp | Zirkularra | Molekula talde heterogenoa |
| 5 | Fungi, Landare, Protista | Ez | 1-200 kbp | Lineala | Molekula talde homogeneoa |
| 6 | Protista | Ez | 1-200 kbp | Lineala | Molekula talde heterogenoa |
Animaliak
[aldatu | aldatu iturburu kodea]Animalia gehienek, bereziki bilateroek, mitokondrio-genoma zirkularra dute. Hala ere, medusozoa eta calkarea kladoen mitokondrioak kromosoma linealak dituzte. [8]
Base-pareei dagokienez, Isarachnanthus nocnus anemonak du animalia ororen genoma mitokondrialik handiena, 80,923 pb-ra heltzen da. [9]
2020ko otsailean, marmokekin zerikusia duen parasito bat aurkitu zen, Henneguya salminicola alegia. Parasito honek ez du ez mitokondriorik ezta genoma mitokondrialik ere, baina baditu mitokondrioarekin zerikusia duten organulutzat jotzen diren egiturak. Gainera, ez ditu arnasketa aerobikoan eta DNAren erreplikazioan eta transkripzioan parte hartzen duten DNA nuklearrarren generik; eta agertzen badira, gene hauek pseudogene moduan agertzen dira. Orokorrean, organismo zelulanitz guztiek arnasketa aerobikoa egiten dute, parasito hau ordea salbuesoen bat da. Izan ere, arnasketa aerobikorik ez duen organismo zelulanitz bakarra da eta oxigenoarekiko mendekotasunik gabe bizi da erabat. [10][11]
Landare eta Onddoak
[aldatu | aldatu iturburu kodea]Hiru genoma mitokondrial mota partekatzen dituzte landare eta onddoek. Lehenengo mota genoma zirkular bat da, intronak ditu (goiko taulan azaldutako 2. genoma mota), eta 19 eta 1000 kbp arteko luzera izan dezake. Bigarren genoma mota zirkularra da (20-1000 kbp inguru), eta plasmidoaren antzeko egitura du (1 kbp) (3. genoma mota). Landare eta onddoetan aurkitu daitekeen azken genoma DNA molekula homogeneoz (5. genoma mota) osatutako genoma lineala da.
DNAmt-ko geneen edukian eta tamainan aldaketa handiak daude onddo eta landareen artean. Hala ere, badirudi eukarioto guztietan agertzen den geneen oinarrizko azpimultzo bat dutela landare eta onddoek (mitokondriorik ez dutenek izan ezik). [6] Zenbait landare-espeziek mitokondrio-genoma izugarriak dituzte, eta Silene conica espeziearen DNAmt-ak 11,300.000 base-pare ditu.[12]
Harrigarria bada ere, DNAmt ikaragarri horiek dituzten espezieak eta DNAmt askoz txikiagoak dituzten espezieak gene-kopuru eta -mota berberak dituzte. [13]
Pepinoaren (Cucumis sativus) mitokondrioko genomak hiru kromosoma zirkular ditu (1556, 84 eta 45 kbp luzerak), eta guztiz edo erabat autonomoak dira erreplikazioari dagokionez. [14]
Protistoak
[aldatu | aldatu iturburu kodea]Protistoen artean askotarito genoma mitokondrialak aurkitzen dira, zehazki, 5 mota ezberdin deskribatu dira. Landareekin partekatzen dituzten 3 genoma motez gain (2, 3 eta 5. motak), badituzte beste 2 mota bereizgarri. Azken horietako bat DNA molekula zirkularren (4. mota) bilduma heterogeneoa da; bestea, berriz, molekula linealen bilduma heterogeneoa (6. mota). 4. zein 6. genoma motak 1 eta 200 kbp bitartekoak dira.
Orain arte sekuentziatutako genoma mitokondrial txikiena Plasmodium falciparum parasitoarena izan da, 5.967 p-koa. [15][16]
Geneen transferentzia endosinbiotikoaren bidez, mitokondrio-genoman kodetutako geneak zelularen genoma nagusira transferitzen dira. Honek azaldu dezake zergatik dituzten mitokondrio-genoma txikiagoak gizakiek (organismo konplexuak izanik) protistek baino (hauek organismo sinpleagoak dira). Azkenean, konplexutasunak gora egin ahala, gene horiek mitokondrioa utzi eta nukleora igarotzen baitira.
Herentzia mitokondriala
[aldatu | aldatu iturburu kodea]Zelula anitzeko organismo gehienetan, DNAmt-a amarengandik heredatzen da. [17]
2018an argitaratutako ikerketa batek aditzera eman du gizakiek DNAmt-a bai gurasoengandik bai amengandik jasotzen dutela eta honek DNAmt-ren heteroplasmia eragiten duela[18]. Heteroplasmia da organuluen genoma bat baino gehiago izatea. Hala ere, komunitate zientifikoak baztertu egiten du hori.
Herentzia amatiarra
[aldatu | aldatu iturburu kodea]Ugalketa sexualean, mitokondrioak amarengandik soilik heredatzen dira; Gehienetan ugaztunen obuluek espermatozoideen mitokondrioak suntsitzen ditu ernalketaren ostean. Gainera, mitokondrioak soilik espermaren isatsean kokatzen dira, elementu hau zelularen mugimendurako erabiltzen da, eta batzuetan ernalketan zehar galtzen da. 1999an jakinarazi zen aitaren espermako mitokondrioak (DNAmt-ak dituztenak) ubikitinaz markatuta daudela, enbrioiaren barruan suntsitzeko[19]. In vitro ernalketa-teknika batzuek, obozito batean esperma injektatzeak bereziki, prozesu hauei eragin diezaiekete.
Mitokondrioko DNA nagusiki herentzia amatiarrekoa izateak aukera ematen die ikertzaile genealogikoei ama-leinua denboran oso atzera arakatzeko. Y DNA kromosomikoa, paternalki jasoa, antzera erabiltzen da historia patrilineala zehazteko. Hori gizakiaren DNA mitokondrialean egiten da, kontrol-eskualde hiperaldakorrak (HVR1 edo HVR2) sekuentziatuz; batzuetan, DNA mitokondrialaren molekula osoa erabiltzen da, DNA genealogikoaren froga gisa. HVR1 eskualdeak, adibidez, 440 base-pare ditu. 440 base-pare horiek beste indibiduo batzuen eskualde analogoekin alderatzen dira (pertsona espezifikoak edo datu-base bateko subjektuak), leinu amatiarra zehazteko. Askotan, alderaketa “Cambridge Reference Sequence”-en bidez egiten da. Ikerlari batzuek otsoen eta etxeko zakurren arteko erlazio matrilinealaren inguruko azterketak argitaratu dituzte[20]. Eva Mitokondrialaren kontzeptua analisi-mota berean oinarritzen da, gizateriaren jatorria ezagutzeko denboran zehar leinua arakatuz.
DNA mitokodrialaren botila-lepoa
[aldatu | aldatu iturburu kodea]Guraso bakarreko herentziari lotuta dauden eta birkonbinazio gutxi edo batere ez duten entitateak Mullerren trinketeari lotzen zaizkio, hots, mutazio kaltegarriak era itzulezinean metatzeari, funtzionaltasuna galdu arte. Animalia-erreinuko mitokondrio populazioak, trinketea ekiditzeko, DNAmt-en botila-lepoa izeneko garapen-prozesuaz baliatzen dira. Botila-lepoak zelulako zorizko prozesuak areagotzen ditu zelulen arteko mutazio kargan aldakortasuna emendatzeko, organismo bat garatu ahala: DNAmt mutantearen proportzio bat duen obozito bakar batek enbrioi bat sortzen du, enbrioiko zelula desberdinek karga mutante desberdinak izango dituztelarik. Zelula-mailan ematen den hautaketak ahalbidetzen du DNAmt-mutante gehien duten zelulak ezabatzea, eta, horren ondorioz, belaunaldien arteko mutazio karga egonkortu edo murriztu egiten da. Berriki egindako meta-azterketa matematiko eta esperimental bat dela eta[21], botila-lepoaren mekanismoa eztabaidapean dago[22] [23][24][25].
Herentzia aitatiarra
[aldatu | aldatu iturburu kodea]Plymouth Rock-eko oiloetan aurkitu da DNA mitokondrialaren herentzia aitatiarra[26]. Ugaztun batzuetan ere herentzia mitokondrial aitatiarreko kasu bakanak babesten ditu ebidentziak. Zehazki, arratoietarako gertaerak daude dokumentatuta[27][28], eta horietan herentziazko mitokondria maskulinoak baztertu egin ziren geroago. Ardi eta behi klonatuetan ere aurkitu da[29] [30]. Giza mitokondrio herentzia kasu arraroak dokumentatu dira[31][32][33]. Kasu horietako asko enbrioi klonatuak edo aita-mitokondria baztertzearen ondorioak dira; hala ere, beste batzuek in vivo herentzia eta iraunkortasuna dokumentatzen dituzte, baina hau soilik laborategian ikusi da.
Donazio mitokondriala
[aldatu | aldatu iturburu kodea]Donazio mitokondrial edo mitokondrio-ordezkapeneko terapia izenez ezagutzen den In vitro ernalketa teknikaren kasuan, enbrioiak eme emailearen DNAmt eta aita-amen DNA nuklearra jasotzen du. Transferentzia bidez aktibatutako obozitoen teknikan, obulu baten nukleoa eme emaile baten obulu baten zitoplasman sartzen da. Emailearen obuluak bere nukleoa galdu du, baina oraindik eme emailearen DNAmt dauka. Obulu hau arraren espermarekin elkartzen da. Prozedura hau genetikoki akastunak diren mitokondrioak dituzten emakumeen kasuan erabiltzen da, era honetara seme-alabek mitokondrio osasuntsuak jasoko dituzte.
Mutazio eta gaixotasunak
[aldatu | aldatu iturburu kodea]DNAmt-ak arnas kateak sortutako oxigeno-espezie erreaktiboekiko (ROS) izan dezaken sentikortasuna eztabaidan dago, bi egituren gertutasunaren ondorioz[34]. DNAmt-ak ez du DNA nuklearrak baino kalte oxidatiborik metatzen[35]. Are gehiago, DNA-n oxidazio kalte batzuk modu eraginkorragoan konpontzen dira mitokondrioetan nukleoan baino[36]. DNAmt-a kromatina nuklearreko proteinak bezain babesleak diruditen proteinekin paketatuta dago[37]. Gainera, mitokondrioek DNAmt osasuntsua mantentzeko mekanismo berezi bat garatu dute; hondatuegiak dauden genomak degradatu eta konponduta edo osorik dauden genomak soilik erreplikatzen dituzte. Mekanismo hori ez da nukleoan aurkitzen[38]. DNAmt-aren mutazioek zenbait proteinen kodean aldaketa izan dezake, eta horrek eragina izan dezake organismoaren metabolismo edota egoera fisikoan.
Neuroendekapenezko gaixotasunak
[aldatu | aldatu iturburu kodea]DNAmt-ri eragindako kaltea handitzea zenbait gaixotasun neurodegeneratiboren ezaugarria da.
Alzheimer gaixotasuna duten pertsonen garunek DNAri kalte oxidatibo handia egiten diote, bai DNAn bai DNAmt-an[39]. Proposatu da mitokondrio zahartuak faktore kritikoa direla Alzheimer gaixotasunaren neurodegenerazioaren jatorrian[40].
Huntingtonen gaixotasunean proteina mutante huntingtinak disfuntzio mitokondriala eragiten du, eta horrek eragiten du elektroi mitokondrialen garraioaren inhibizioa, oxigeno-espezie erreaktiboen maila handiagoa eta oxidazio-estresa areagotzea[41]. Huntingtina proteina mutanteak kalte oxidatiboa eragiten dio DNAmt-ri, bai eta DNA nuklearrari ere, eta azken horrek Huntington-en gaixotasunaren patologian laguntzen du[42].
DNAren oxidazio-produktua 8-oxoguanina (8-oxoG) da eta DNAren oxidazio-kaltearen markatzaile nabarmena da. Alboko esklerosi amiotrofikoa (ELA) duten pertsonengan 8-oxoG DNAren kalteak, neurona motore espinalen DNAmt-koak, konpondu ohi dituzten entzimak kaltetuta daude[43]. Hala, neurona motorren DNAmt-ri egindako oxidazio-kaltea faktore esanguratsua izan daiteke ELAren etiologian.
DNAmt-en oinarriaren konposizioaren eta animalia-bizitzaren arteko korrelazioa
[aldatu | aldatu iturburu kodea]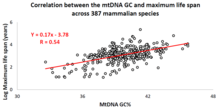
Azken hamarkadan Vadim Fraifeld irakasleak zuzendutako Israelgo ikerketa-talde batek frogatu du korrelazio sendo eta esanguratsuak daudela DNAmt-en oinarriaren konposizioaren eta animalia-espezie bakoitzaren bizitza maximo espezifikoaren artean[44][45][45]. Bere lanean frogatu zen moduan, DNAmt guanina + zitosina (% GC) gehiago animalia-espezieen artean bizi-itxaropen maximo handiagoarekin lotzen da[45].
Erabilera biologia ebolutiboan eta biologia sistematikoan
[aldatu | aldatu iturburu kodea]DNAmt-a organismo eukariotikoetan aurkitzen da, eta mitokondrioetan arnasketa zelularraren zeregin kritikoa egiten du. Hala ere, eraginkortasun gutxiagoko DNAren konponketa baten ondorioz (DNA nuklearrarekin konparatuta), mutazio-tasa nahiko handia du DNAmt-a (baina motela beste DNA eskualde batzuekin alderatuta, hala nola mikrosateliteekin), eta hori baliagarria da organismoen eboluzio-erlazioak aztertzeko - filogenia -. Biologoek espezie desberdinen arteko DNAm-en sekuentziak zehaztu eta gero konpara ditzakete, eta konparazioak erabil ditzakete aztertutako espezieentzat zuhaitz ebolutibo bat eraikitzeko.
Adibidez, gene nuklear gehienak ia berdinak dira gizakien eta txinpantzeen artean, baina mitokondrio-genomak % 9,8 ezberdintasuna daukate. Bestalde, giza mitokondrio-genomak eta gorilarenak % 11,8ean desberdinak dira. Honen bitartez ondorioztatzen da antz handiagoa daukagula txinpantzeekin gorilekin baino[46].
Historia
[aldatu | aldatu iturburu kodea]DNA mitokondriala 1960ko hamarkadan aurkitu zuten Margit M. K. Nass eta Sylvan Nass-ek, mikroskopia elektronikoaren bidez[47], eta Ellen Haslbrunnerrek, Hans Tuppyk eta Gottfried Schatzek, saiakuntza biokimikoak eginez oso purifikatutako frakzio mitokondrialen gainean[48].
Mitokondrio-sekuentzien datu-baseak
[aldatu | aldatu iturburu kodea]Genoma mitokondrialaren sekuentziak eta bestelako informazioak biltzeko datu-base espezializatu batzuk sortu dira. Gehienak sekuentzia-datuetan oinarritzen diren arren, batzuek informazio filogenetikoa edo funtzionala dute.
- AmtDB: gizakien arbasoen mitokondrio-genoma datu-basea[49].
- InterMitoBase: giza mitokondrioentzako proteina-proteina elkarrekintzak aztertzeko plataforma[50].
- MitoBreak: mitokondrioko DNAren ebakitze-puntuen datu-basea[51].
- MitoFish eta MitoAnnotator: arrainen genoma mitokondrialaren datu-basea[52][53].
- MitoRes: nukleoan kodetutako gene mitokondrialen baliabide bat eta haren produktuak metazooetan[54].
DNAmt-aren fenotipoen elkarketaren datu-baseak
[aldatu | aldatu iturburu kodea]Genoma osoan egiten diren elkartze-azterketek DNAmt-eko gene-elkarteak eta horien mutazioak fenotipoekin erakuts ditzakete, bizi-itxaropena eta gaixotasun-arriskuak barne. 2021ean mitokondrioko DNAren elkartze-azterlan handiena, Erresuma Batuko Biobankuan oinarritua, genoma osoan, 260 elkarte berri sortu ziren fenotipoekin, bizi-itxaropena eta gaixotasun-arriskuak barne, adibidez 2. motako diabetesa[55][56].
Mitokondrio-mutazioen datu-baseak
[aldatu | aldatu iturburu kodea]Zenbait datu-base espezializatuk polimorfismoak eta mutazioak dituzte giza mitokondrioko DNAn eta, horrekin batera, patogenotasunaren ebaluazioa egiten dute.
- MitImpact: Aurrez kalkulatutako patogenotasunaren iragarpenen bilduma bat da. Kalkulu horietarako aldaketa nukleotiko batzuk erabiltzen dira eta aldaketa nukleotiko horiek guztiek giza proteina mitokondrialen gene kodetzaileen sinonimoak ez diren ordezkapenak eragiten dituzten[57].
- MITOMAP: Polimorfismo eta mutazioen bilduma giza mitokondrioko DNAn.
Erreferentziak
[aldatu | aldatu iturburu kodea]- ↑ «news-from-human-rights-watch-no1g-dangerous-dealings-changes-to-us-military-assistance-after-september-11-february-2002-15-pp» Human Rights Documents online (Noiz kontsultatua: 2021-10-29).
- ↑ (Ingelesez) Anderson, S.; Bankier, A. T.; Barrell, B. G.; de Bruijn, M. H. L.; Coulson, A. R.; Drouin, J.; Eperon, I. C.; Nierlich, D. P. et al.. (1981-04). «Sequence and organization of the human mitochondrial genome» Nature 290 (5806): 457–465. doi:. ISSN 0028-0836. (Noiz kontsultatua: 2021-10-29).
- ↑ (Ingelesez) Boursot, P; Bonhomme, F. (1986). «Génétique et évolution du génome mitochondrial des Métazoaires» Genetics Selection Evolution 18 (1): 73. doi:. ISSN 1297-9686. PMID 22879234. PMC PMC2713894. (Noiz kontsultatua: 2021-10-29).
- ↑ (Ingelesez) Hassanin, Alexandre; An, Junghwa; Ropiquet, Anne; Nguyen, Trung Thanh; Couloux, Arnaud. (2013-03). «Combining multiple autosomal introns for studying shallow phylogeny and taxonomy of Laurasiatherian mammals: Application to the tribe Bovini (Cetartiodactyla, Bovidae)» Molecular Phylogenetics and Evolution 66 (3): 766–775. doi:. (Noiz kontsultatua: 2021-10-29).
- ↑ (Ingelesez) Delsuc, Frédéric; Stanhope, Michael J; Douzery, Emmanuel J.P. (2003-08). «Molecular systematics of armadillos (Xenarthra, Dasypodidae): contribution of maximum likelihood and Bayesian analyses of mitochondrial and nuclear genes» Molecular Phylogenetics and Evolution 28 (2): 261–275. doi:. (Noiz kontsultatua: 2021-10-29).
- ↑ a b Johnston, Iain G.; Williams, Ben P.. (2016-02). «Evolutionary Inference across Eukaryotes Identifies Specific Pressures Favoring Mitochondrial Gene Retention» Cell Systems 2 (2): 101–111. doi:. ISSN 2405-4712. (Noiz kontsultatua: 2021-10-29).
- ↑ (Ingelesez) Kolesnikov, A. A.; Gerasimov, E. S.. (2012-12). «Diversity of mitochondrial genome organization» Biochemistry (Moscow) 77 (13): 1424–1435. doi:. ISSN 0006-2979. (Noiz kontsultatua: 2021-10-29).
- ↑ (Ingelesez) Lavrov, Dennis V.; Pett, Walker. (2016-09). «Animal Mitochondrial DNA as We Do Not Know It: mt-Genome Organization and Evolution in Nonbilaterian Lineages» Genome Biology and Evolution 8 (9): 2896–2913. doi:. ISSN 1759-6653. PMID 27557826. PMC PMC5633667. (Noiz kontsultatua: 2021-10-29).
- ↑ (Ingelesez) Stampar, Sérgio N.; Broe, Michael B.; Macrander, Jason; Reitzel, Adam M.; Brugler, Mercer R.; Daly, Marymegan. (2019-04-15). «Linear Mitochondrial Genome in Anthozoa (Cnidaria): A Case Study in Ceriantharia» Scientific Reports 9 (1): 6094. doi:. ISSN 2045-2322. PMID 30988357. PMC PMC6465557. (Noiz kontsultatua: 2021-10-29).
- ↑ (Ingelesez) Yahalomi, Dayana; Atkinson, Stephen D.; Neuhof, Moran; Chang, E. Sally; Philippe, Hervé; Cartwright, Paulyn; Bartholomew, Jerri L.; Huchon, Dorothée. (2020-03-10). «A cnidarian parasite of salmon (Myxozoa: Henneguya) lacks a mitochondrial genome» Proceedings of the National Academy of Sciences 117 (10): 5358–5363. doi:. ISSN 0027-8424. PMID 32094163. PMC PMC7071853. (Noiz kontsultatua: 2021-10-29).
- ↑ STARR, J.R.. (1981). «CLIMATE AND THE NEED FOR HOUSING» Environmental Aspects of Housing for Animal Production (Elsevier): 19–35. (Noiz kontsultatua: 2021-10-29).
- ↑ (Ingelesez) Sloan, Daniel B.; Alverson, Andrew J.; Chuckalovcak, John P.; Wu, Martin; McCauley, David E.; Palmer, Jeffrey D.; Taylor, Douglas R.. (2012-01-17). «Rapid Evolution of Enormous, Multichromosomal Genomes in Flowering Plant Mitochondria with Exceptionally High Mutation Rates» PLOS Biology 10 (1): e1001241. doi:. ISSN 1545-7885. PMID 22272183. PMC PMC3260318. (Noiz kontsultatua: 2021-10-29).
- ↑ Ward, Bernard L.; Anderson, Robert S.; Bendich, Arnold J.. (1981-09). «The mitochondrial genome is large and variable in a family of plants (Cucurbitaceae)» Cell 25 (3): 793–803. doi:. ISSN 0092-8674. (Noiz kontsultatua: 2021-10-29).
- ↑ Alverson, Andrew J; Rice, Danny W; Dickinson, Stephanie; Barry, Kerrie; Palmer, Jeffrey D. (2011-07). «Origins and Recombination of the Bacterial-Sized Multichromosomal Mitochondrial Genome of Cucumber» The Plant Cell 23 (7): 2499–2513. doi:. ISSN 1040-4651. PMID 21742987. PMC PMC3226218. (Noiz kontsultatua: 2021-10-29).
- ↑ Goldstein, Amy. (2016). «MPV17-Related Hepatocerebral Mitochondrial DNA (mtDNA) Depletion Syndrome» Mitochondrial Case Studies (Elsevier): 179–185. (Noiz kontsultatua: 2021-10-29).
- ↑ (Ingelesez) Tyagi, Suchi; Pande, Veena; Das, Aparup. (2014-02-19). «Whole Mitochondrial Genome Sequence of an Indian Plasmodium falciparum Field Isolate» The Korean Journal of Parasitology 52 (1): 99–103. doi:. ISSN 0023-4001. PMID 24623891. PMC PMC3949004. (Noiz kontsultatua: 2021-10-29).
- ↑ (Ingelesez) Wolff, J. N.; Gemmell, N. J.. (2008-11). «Lost in the zygote: the dilution of paternal mtDNA upon fertilization» Heredity 101 (5): 429–434. doi:. ISSN 1365-2540. (Noiz kontsultatua: 2021-10-29).
- ↑ (Ingelesez) Luo, Shiyu; Valencia, C. Alexander; Zhang, Jinglan; Lee, Ni-Chung; Slone, Jesse; Gui, Baoheng; Wang, Xinjian; Li, Zhuo et al.. (2018-12-18). «Biparental Inheritance of Mitochondrial DNA in Humans» Proceedings of the National Academy of Sciences 115 (51): 13039–13044. doi:. ISSN 0027-8424. PMID 30478036. PMC PMC6304937. (Noiz kontsultatua: 2021-10-29).
- ↑ Travis, John. (2000-01-01). «Mom's Eggs Execute Dad's Mitochondria» Science News 157 (1): 5. doi:. (Noiz kontsultatua: 2021-11-02).
- ↑ (Ingelesez) Vilà, Carles; Savolainen, Peter; Maldonado, Jesús E.; Amorim, Isabel R.; Rice, John E.; Honeycutt, Rodney L.; Crandall, Keith A.; Lundeberg, Joakim et al.. (1997-06-13). «Multiple and Ancient Origins of the Domestic Dog» Science 276 (5319): 1687–1689. doi:. ISSN 0036-8075. (Noiz kontsultatua: 2021-11-02).
- ↑ (Ingelesez) Johnston, Iain G; Burgstaller, Joerg P; Havlicek, Vitezslav; Kolbe, Thomas; Rülicke, Thomas; Brem, Gottfried; Poulton, Jo; Jones, Nick S. (2015-06-02). «Stochastic modelling, Bayesian inference, and new in vivo measurements elucidate the debated mtDNA bottleneck mechanism» eLife 4: e07464. doi:. ISSN 2050-084X. PMID 26035426. PMC PMC4486817. (Noiz kontsultatua: 2021-11-02).
- ↑ (Ingelesez) Wolff, Jonci N.; White, Daniel J.; Woodhams, Michael; White, Helen E.; Gemmell, Neil J.. (2011-05-31). Gilbert, M. Thomas P. ed. «The Strength and Timing of the Mitochondrial Bottleneck in Salmon Suggests a Conserved Mechanism in Vertebrates» PLoS ONE 6 (5): e20522. doi:. ISSN 1932-6203. PMID 21655224. PMC PMC3105079. (Noiz kontsultatua: 2021-11-02).
- ↑ (Ingelesez) Cree, Lynsey M; Samuels, David C; de Sousa Lopes, Susana Chuva; Rajasimha, Harsha Karur; Wonnapinij, Passorn; Mann, Jeffrey R; Dahl, Hans-Henrik M; Chinnery, Patrick F. (2008-02). «A reduction of mitochondrial DNA molecules during embryogenesis explains the rapid segregation of genotypes» Nature Genetics 40 (2): 249–254. doi:. ISSN 1061-4036. (Noiz kontsultatua: 2021-11-02).
- ↑ (Ingelesez) Cao, Liqin; Shitara, Hiroshi; Horii, Takuro; Nagao, Yasumitsu; Imai, Hiroshi; Abe, Kuniya; Hara, Takahiko; Hayashi, Jun-Ichi et al.. (2007-03). «The mitochondrial bottleneck occurs without reduction of mtDNA content in female mouse germ cells» Nature Genetics 39 (3): 386–390. doi:. ISSN 1061-4036. (Noiz kontsultatua: 2021-11-02).
- ↑ (Ingelesez) Wai, Timothy; Teoli, Daniella; Shoubridge, Eric A. (2008-12). «The mitochondrial DNA genetic bottleneck results from replication of a subpopulation of genomes» Nature Genetics 40 (12): 1484–1488. doi:. ISSN 1061-4036. (Noiz kontsultatua: 2021-11-02).
- ↑ (Ingelesez) Alexander, Michelle; Ho, Simon Y. W.; Molak, Martyna; Barnett, Ross; Carlborg, Örjan; Dorshorst, Ben; Honaker, Christa; Besnier, Francois et al.. (2015-10). «Mitogenomic analysis of a 50-generation chicken pedigree reveals a rapid rate of mitochondrial evolution and evidence for paternal mtDNA inheritance» Biology Letters 11 (10): 20150561. doi:. ISSN 1744-9561. PMID 26510672. PMC PMC4650172. (Noiz kontsultatua: 2021-11-02).
- ↑ (Ingelesez) Gyllensten, Ulf; Wharton, Dan; Josefsson, Agneta; Wilson, Allan C.. (1991-07). «Paternal inheritance of mitochondrial DNA in mice» Nature 352 (6332): 255–257. doi:. ISSN 0028-0836. (Noiz kontsultatua: 2021-11-02).
- ↑ Shitara, Hiroshi; Hayashi, Jun-Ichi; Takahama, Sumiyo; Kaneda, Hideki; Yonekawa, Hiromichi. (1998-02-01). «Maternal Inheritance of Mouse mtDNA in Interspecific Hybrids: Segregation of the Leaked Paternal mtDNA Followed by the Prevention of Subsequent Paternal Leakage» Genetics 148 (2): 851–857. doi:. ISSN 1943-2631. (Noiz kontsultatua: 2021-11-02).
- ↑ (Ingelesez) Zhao, X.; Li, N.; Guo, W.; Hu, X.; Liu, Z.; Gong, G.; Wang, A.; Feng, J. et al.. (2004-10). «Further evidence for paternal inheritance of mitochondrial DNA in the sheep (Ovis aries)» Heredity 93 (4): 399–403. doi:. ISSN 1365-2540. (Noiz kontsultatua: 2021-11-02).
- ↑ (Ingelesez) Steinborn, Ralf; Zakhartchenko, Valeri; Jelyazkov, Jivko; Klein, Dieter; Wolf, Eckhard; Müller, Mathias; Brem, Gottfried. (1998). «Composition of parental mitochondrial DNA in cloned bovine embryos» FEBS Letters 426 (3): 352–356. doi:. ISSN 1873-3468. (Noiz kontsultatua: 2021-11-02).
- ↑ Singh, Abhishek Narain. (2018-05-16). «Customized biomedical informatics» Big Data Analytics 3 (1): 6. doi:. ISSN 2058-6345. (Noiz kontsultatua: 2021-11-02).
- ↑ Schwartz, Marianne; Vissing, John. (2002-08-22). «Paternal inheritance of mitochondrial DNA» The New England Journal of Medicine 347 (8): 576–580. doi:. ISSN 1533-4406. PMID 12192017. (Noiz kontsultatua: 2021-11-02).
- ↑ (Ingelesez) Luo, Shiyu; Valencia, C. Alexander; Zhang, Jinglan; Lee, Ni-Chung; Slone, Jesse; Gui, Baoheng; Wang, Xinjian; Li, Zhuo et al.. (2018-12-18). «Biparental Inheritance of Mitochondrial DNA in Humans» Proceedings of the National Academy of Sciences 115 (51): 13039–13044. doi:. ISSN 0027-8424. PMID 30478036. PMC PMC6304937. (Noiz kontsultatua: 2021-11-02).
- ↑ (Ingelesez) Alexeyev, Mikhail F.. (2009). «Is there more to aging than mitochondrial DNA and reactive oxygen species?» The FEBS Journal 276 (20): 5768–5787. doi:. ISSN 1742-4658. PMID 19796285. PMC PMC3097520. (Noiz kontsultatua: 2021-11-02).
- ↑ Anson, R. M.; Hudson, E.; Bohr, V. A.. (2000-02). «Mitochondrial endogenous oxidative damage has been overestimated» FASEB journal: official publication of the Federation of American Societies for Experimental Biology 14 (2): 355–360. doi:. ISSN 0892-6638. PMID 10657991. (Noiz kontsultatua: 2021-11-02).
- ↑ (Ingelesez) Thorslund, Tina; Sunesen, Morten; Bohr, Vilhelm A.; Stevnsner, Tinna. (2002-04-29). «Repair of 8-oxoG is slower in endogenous nuclear genes than in mitochondrial DNA and is without strand bias» DNA Repair 1 (4): 261–273. doi:. ISSN 1568-7864. (Noiz kontsultatua: 2021-11-02).
- ↑ Guliaeva, N. A.; Kuznetsova, E. A.; Gaziev, A. I.. (2006-07). «[Proteins associated with mitochondrial DNA protect it against the action of X-rays and hydrogen peroxide»] Biofizika 51 (4): 692–697. ISSN 0006-3029. PMID 16909848. (Noiz kontsultatua: 2021-11-02).
- ↑ (Ingelesez) Alexeyev, Mikhail; Shokolenko, Inna; Wilson, Glenn; LeDoux, Susan. (2013-05-01). «The Maintenance of Mitochondrial DNA Integrity—Critical Analysis and Update» Cold Spring Harbor Perspectives in Biology 5 (5): a012641. doi:. ISSN 1943-0264. PMID 23637283. PMC PMC3632056. (Noiz kontsultatua: 2021-11-02).
- ↑ Wang, J.; Xiong, S.; Xie, C.; Markesbery, W. R.; Lovell, M. A.. (2005-05). «Increased oxidative damage in nuclear and mitochondrial DNA in Alzheimer's disease» Journal of Neurochemistry 93 (4): 953–962. doi:. ISSN 0022-3042. PMID 15857398. (Noiz kontsultatua: 2021-11-02).
- ↑ Bonda, David J.; Wang, Xinglong; Lee, Hyoung-Gon; Smith, Mark A.; Perry, George; Zhu, Xiongwei. (2014-04). «Neuronal failure in Alzheimer's disease: a view through the oxidative stress looking-glass» Neuroscience Bulletin 30 (2): 243–252. doi:. ISSN 1995-8218. PMID 24733654. PMC 4097013. (Noiz kontsultatua: 2021-11-02).
- ↑ Liu, Zewen; Zhou, Tingyang; Ziegler, Alexander C.; Dimitrion, Peter; Zuo, Li. (2017). «Oxidative Stress in Neurodegenerative Diseases: From Molecular Mechanisms to Clinical Applications» Oxidative Medicine and Cellular Longevity 2017: 2525967. doi:. ISSN 1942-0994. PMID 28785371. PMC 5529664. (Noiz kontsultatua: 2021-11-02).
- ↑ Ayala-Peña, Sylvette. (2013-09). «Role of oxidative DNA damage in mitochondrial dysfunction and Huntington's disease pathogenesis» Free Radical Biology & Medicine 62: 102–110. doi:. ISSN 1873-4596. PMID 23602907. PMC 3722255. (Noiz kontsultatua: 2021-11-02).
- ↑ Kikuchi, Hitoshi; Furuta, Akiko; Nishioka, Ken-ichi; Suzuki, Satoshi O.; Nakabeppu, Yusaku; Iwaki, Toru. (2002-04). «Impairment of mitochondrial DNA repair enzymes against accumulation of 8-oxo-guanine in the spinal motor neurons of amyotrophic lateral sclerosis» Acta Neuropathologica 103 (4): 408–414. doi:. ISSN 0001-6322. PMID 11904761. (Noiz kontsultatua: 2021-11-02).
- ↑ Lehmann, Gilad; Budovsky, Arie; Muradian, Khachik K.; Fraifeld, Vadim E.. (2006-06-01). «Mitochondrial Genome Anatomy and Species-Specific Lifespan» Rejuvenation Research 9 (2): 223–226. doi:. ISSN 1549-1684. (Noiz kontsultatua: 2021-11-02).
- ↑ a b c Lehmann, Gilad; Segal, Elena; Muradian, Khachik K.; Fraifeld, Vadim E.. (2008-04-28). «Do Mitochondrial DNA and Metabolic Rate Complement Each Other in Determination of the Mammalian Maximum Longevity?» Rejuvenation Research 11 (2): 409–417. doi:. ISSN 1549-1684. (Noiz kontsultatua: 2021-11-02).
- ↑ Xu, X.; Arnason, U.. (1996-05). «A complete sequence of the mitochondrial genome of the western lowland gorilla» Molecular Biology and Evolution 13 (5): 691–698. doi:. ISSN 0737-4038. PMID 8676744. (Noiz kontsultatua: 2021-11-02).
- ↑ Nass, Margit M. K.; Nass, Sylvan. (1963-12-01). «INTRAMITOCHONDRIAL FIBERS WITH DNA CHARACTERISTICS» The Journal of Cell Biology 19 (3): 593–611. ISSN 0021-9525. PMID 14086138. PMC 2106331. (Noiz kontsultatua: 2021-11-02).
- ↑ Schatz, G.; Haslbrunner, E.; Tuppy, H.. (1964-03-09). «DEOXYRIBONUCLEIC ACID ASSOCIATED WITH YEAST MITOCHONDRIA» Biochemical and Biophysical Research Communications 15 (2): 127–132. doi:. ISSN 0006-291X. PMID 26410904. (Noiz kontsultatua: 2021-11-02).
- ↑ Ehler, Edvard; Novotný, Jirí; Juras, Anna; Chylenski, Maciej; Moravcík, Ondrej; Paces, Jan. (2019-01-08). «AmtDB: a database of ancient human mitochondrial genomes» Nucleic Acids Research 47 (D1): D29–D32. doi:. ISSN 1362-4962. PMID 30247677. PMC 6324066. (Noiz kontsultatua: 2021-11-02).
- ↑ Gu, Zuguang; Li, Jie; Gao, Song; Gong, Ming; Wang, Junling; Xu, Hua; Zhang, Chenyu; Wang, Jin. (2011-06-30). «InterMitoBase: An annotated database and analysis platform of protein-protein interactions for human mitochondria» BMC Genomics 12 (1): 335. doi:. ISSN 1471-2164. PMID 21718467. PMC PMC3142533. (Noiz kontsultatua: 2021-11-02).
- ↑ Damas, Joana; Carneiro, João; Amorim, António; Pereira, Filipe. (2014-01). «MitoBreak: the mitochondrial DNA breakpoints database» Nucleic Acids Research 42 (Database issue): D1261–1268. doi:. ISSN 1362-4962. PMID 24170808. PMC 3965124. (Noiz kontsultatua: 2021-11-02).
- ↑ Iwasaki, Wataru; Fukunaga, Tsukasa; Isagozawa, Ryota; Yamada, Koichiro; Maeda, Yasunobu; Satoh, Takashi P.; Sado, Tetsuya; Mabuchi, Kohji et al.. (2013-11). «MitoFish and MitoAnnotator: a mitochondrial genome database of fish with an accurate and automatic annotation pipeline» Molecular Biology and Evolution 30 (11): 2531–2540. doi:. ISSN 1537-1719. PMID 23955518. PMC 3808866. (Noiz kontsultatua: 2021-11-02).
- ↑ Cawthorn, D.; Steinman, H.; WITTHUHN, R. CORLI. (2011). «Establishment of a mitochondrial DNA sequence database for the identification of fish species commercially available in South Africa» Molecular ecology resources doi:. (Noiz kontsultatua: 2021-11-02).
- ↑ Catalano, Domenico; Licciulli, Flavio; Turi, Antonio; Grillo, Giorgio; Saccone, Cecilia; D'Elia, Domenica. (2006-01-24). «MitoRes: a resource of nuclear-encoded mitochondrial genes and their products in Metazoa» BMC Bioinformatics 7: 36. doi:. ISSN 1471-2105. PMID 16433928. PMC 1395343. (Noiz kontsultatua: 2021-11-02).
- ↑ «Mothers can influence offspring's height, lifespan and disease risk through mitochondria» web.archive.org 2021-06-14 (Noiz kontsultatua: 2021-11-02).
- ↑ «An atlas of mitochondrial DNA genotype–phenotype associations in the UK Biobank | Nature Genetics» web.archive.org 2021-06-13 (Noiz kontsultatua: 2021-11-02).
- ↑ Mazza, Tommaso. «MitImpact 3D - IRCCS-CSS Bioinformatics lab» mitimpact.css-mendel.it (Noiz kontsultatua: 2021-11-02).
